相关系数矩阵怎么做(在Python中创建相关系数矩阵的6种方法)
更新时间:2024-09-22 10:20:34
相关系数矩阵(Correlation matrix)是数据分析的基本工具。它们让我们了解不同的变量是如何相互关联的。在Python中,有很多个方法可以计算相关系数矩阵,今天我们来对这些方法进行一个总结
Pandas
Pandas的DataFrame对象可以使用corr方法直接创建相关矩阵。由于数据科学领域的大多数人都在使用Pandas来获取数据,因此这通常是检查数据相关性的最快、最简单的方法之一。
import pandas as pd
import seaborn as sns
data = sns.load_dataset('mpg')
correlation_matrix = data.corr(numeric_only=True)
correlation_matrix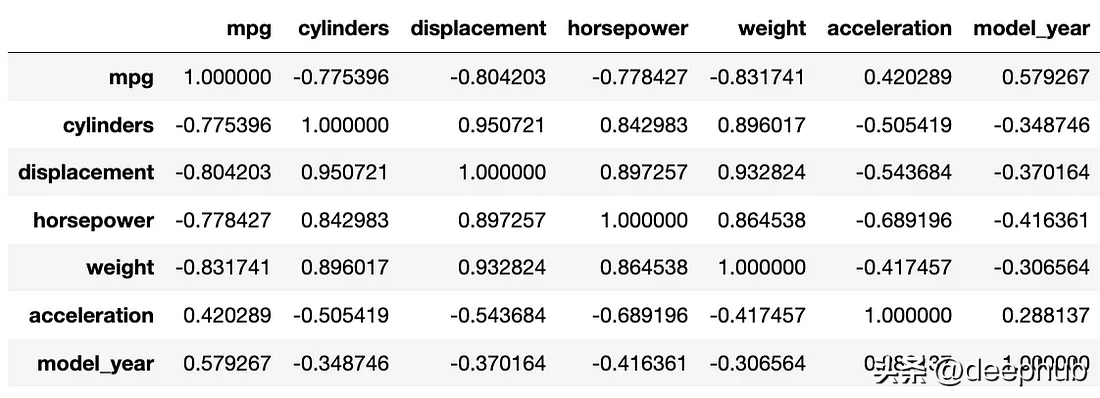
如果你是统计和分析相关工作的,你可能会问" p值在哪里?",在最后我们会有介绍
Numpy
Numpy也包含了相关系数矩阵的计算函数,我们可以直接调用,但是因为返回的是ndarray,所以看起来没有pandas那么清晰。
import numpy as np
from sklearn.datasets import load_iris
iris = load_iris()
np.corrcoef(iris["data"])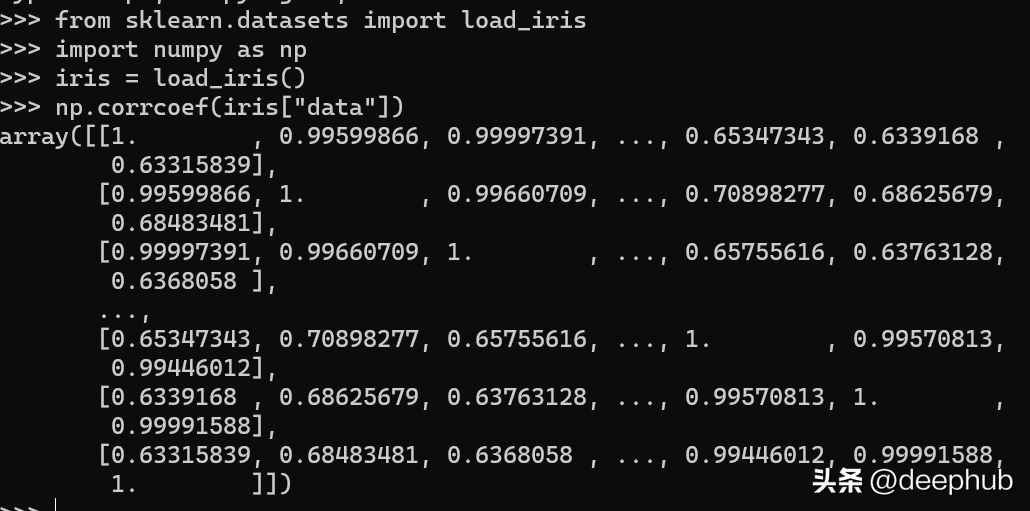
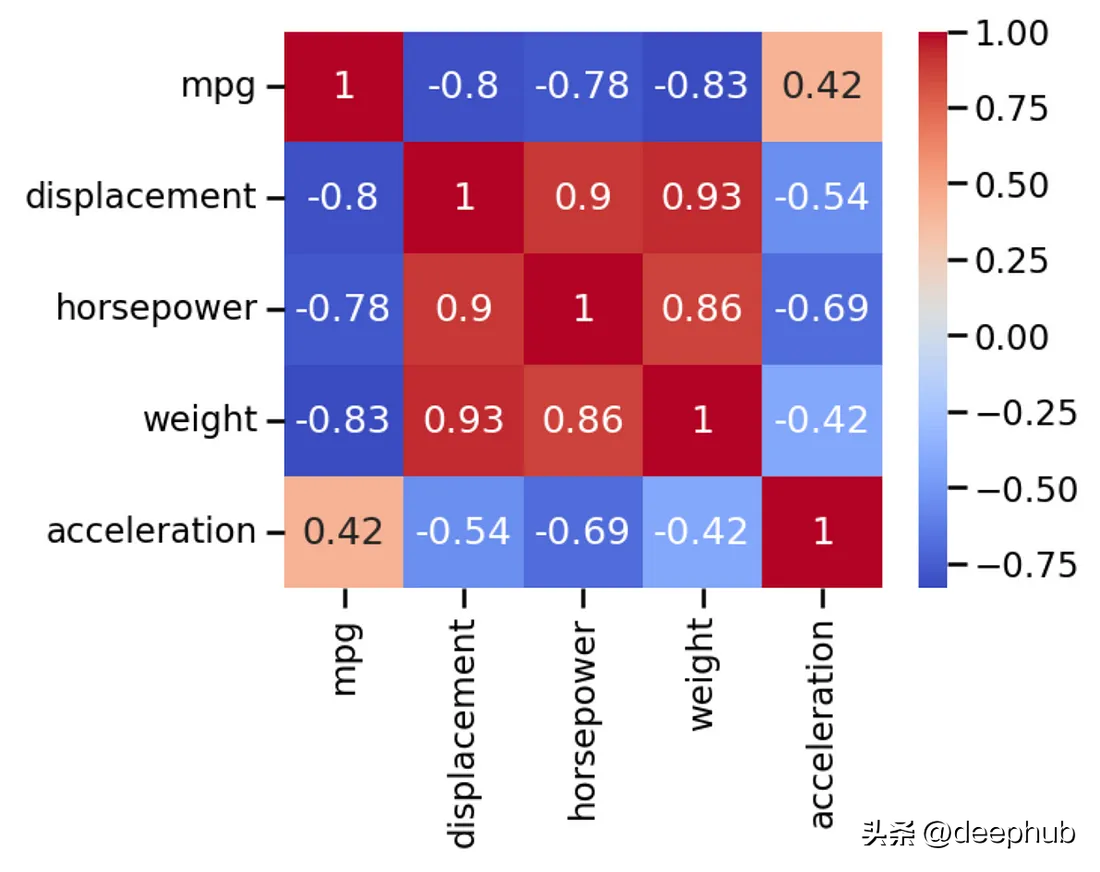
为了更好的可视化,我们可以直接将其传递给sns.heatmap()函数。
import seaborn as sns
data = sns.load_dataset('mpg')
correlation_matrix = data.corr()
sns.heatmap(data.corr(),
annot=True,
cmap='coolwarm')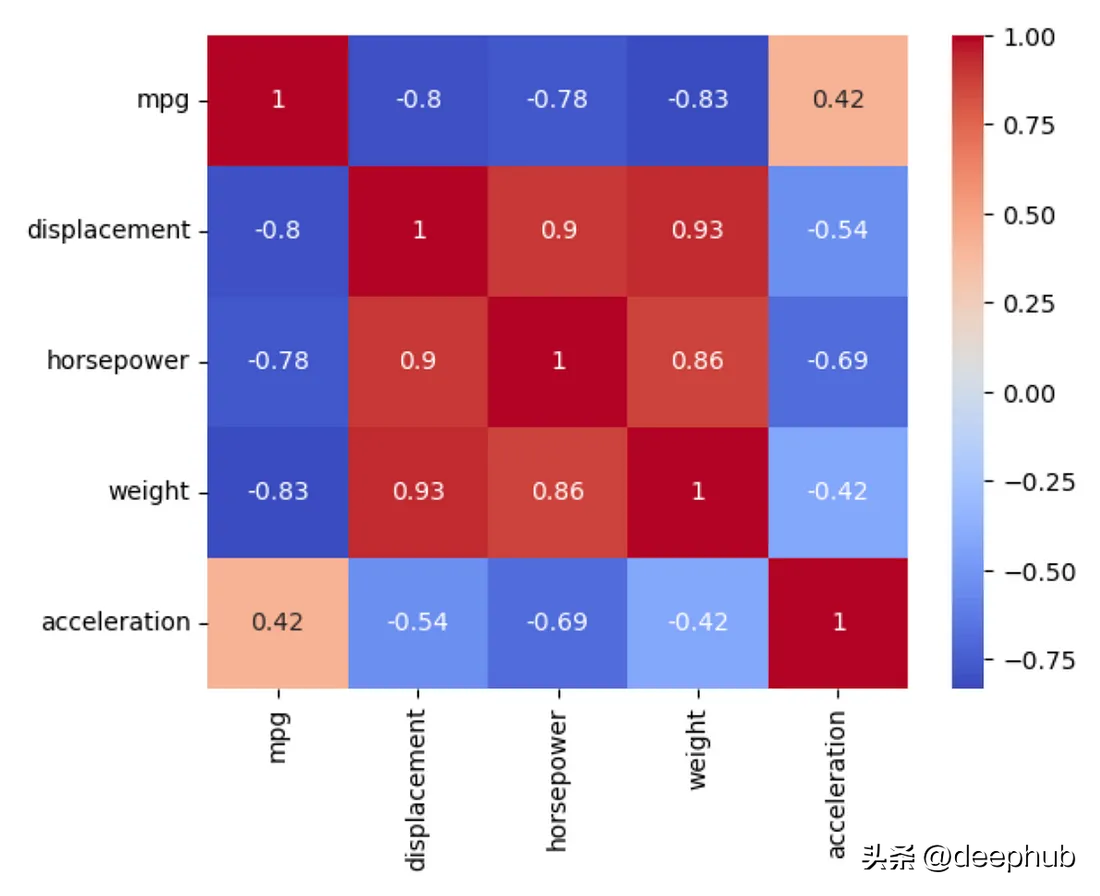
annot=True这个参数可以输出一些额外的有用信息。一个常见hack是使用sns.set_context('talk')来获得额外的可读输出。
这个设置是为了生成幻灯片演示的图像,它能帮助我们更好地阅读(更大的字体)。
Statsmodels
Statsmodels这个统计分析库也是肯定可以的
import statsmodels.api as sm
correlation_matrix = sm.graphics.plot_corr(
data.corr(),
xnames=data.columns.tolist())
plotly
默认情况下plotly这个结果是如何从左下到右上运行对角线1.0的。这种行为与大多数其他工具相反,所以如果你使用plotly需要特别注意
import plotly.offline as pyo
pyo.init_notebook_mode(connected=True)
import plotly.figure_factory as ff
correlation_matrix = data.corr()
fig = ff.create_annotated_heatmap(
z=correlation_matrix.values,
x=list(correlation_matrix.columns),
y=list(correlation_matrix.index),
colorscale='Blues')
fig.show()
Pandas + Matplotlib更好的可视化
这个结果也可以直接使用用sns.pairplot(data),两种方法产生的图差不多,但是seaborn只需要一句话
sns.pairplot(df[['mpg','weight','horsepower','acceleration']])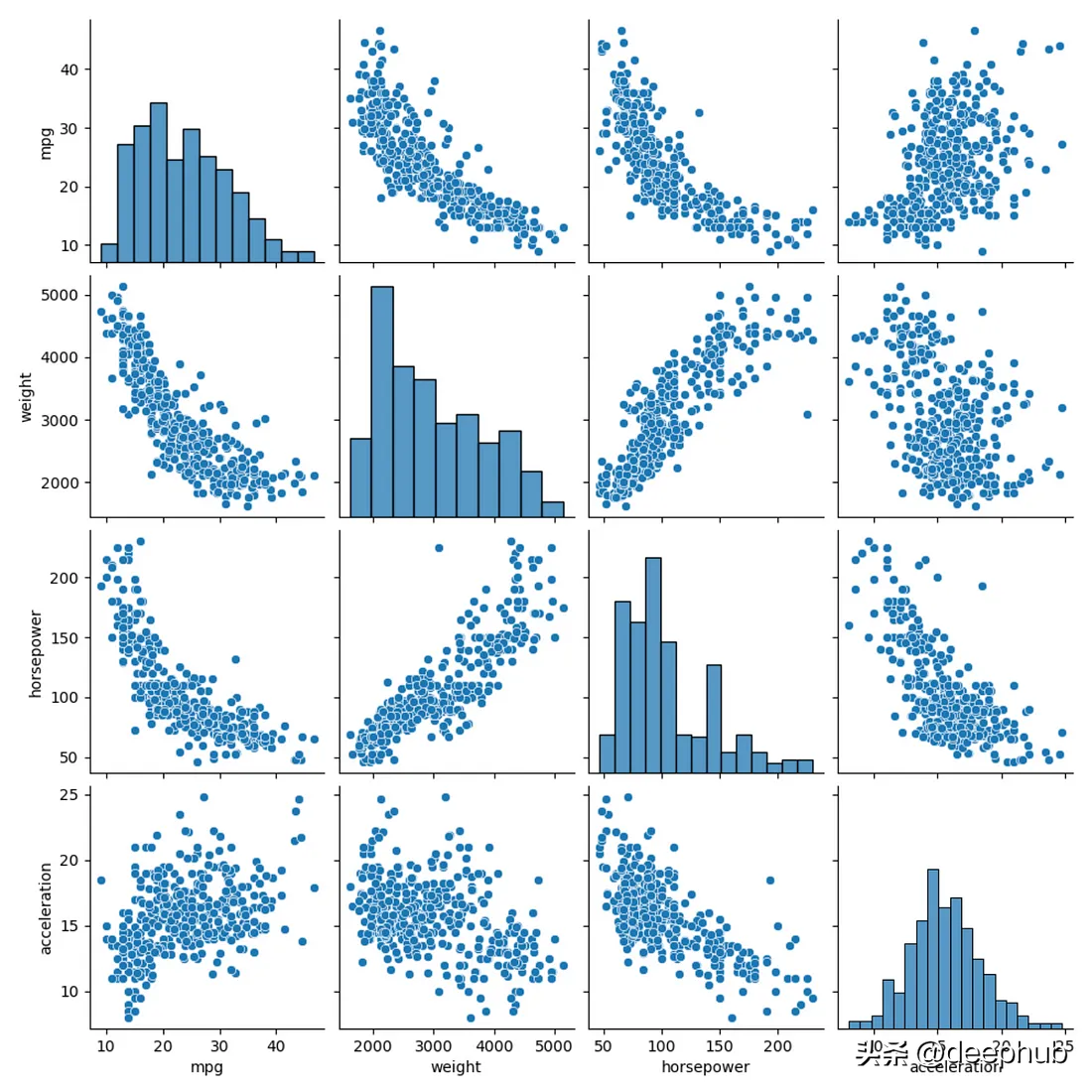
所以我们这里介绍如何使用Matplotlib来实现
import matplotlib.pyplot as plt
pd.plotting.scatter_matrix(
data, alpha=0.2,
figsize=(6, 6),
diagonal='hist')
plt.show()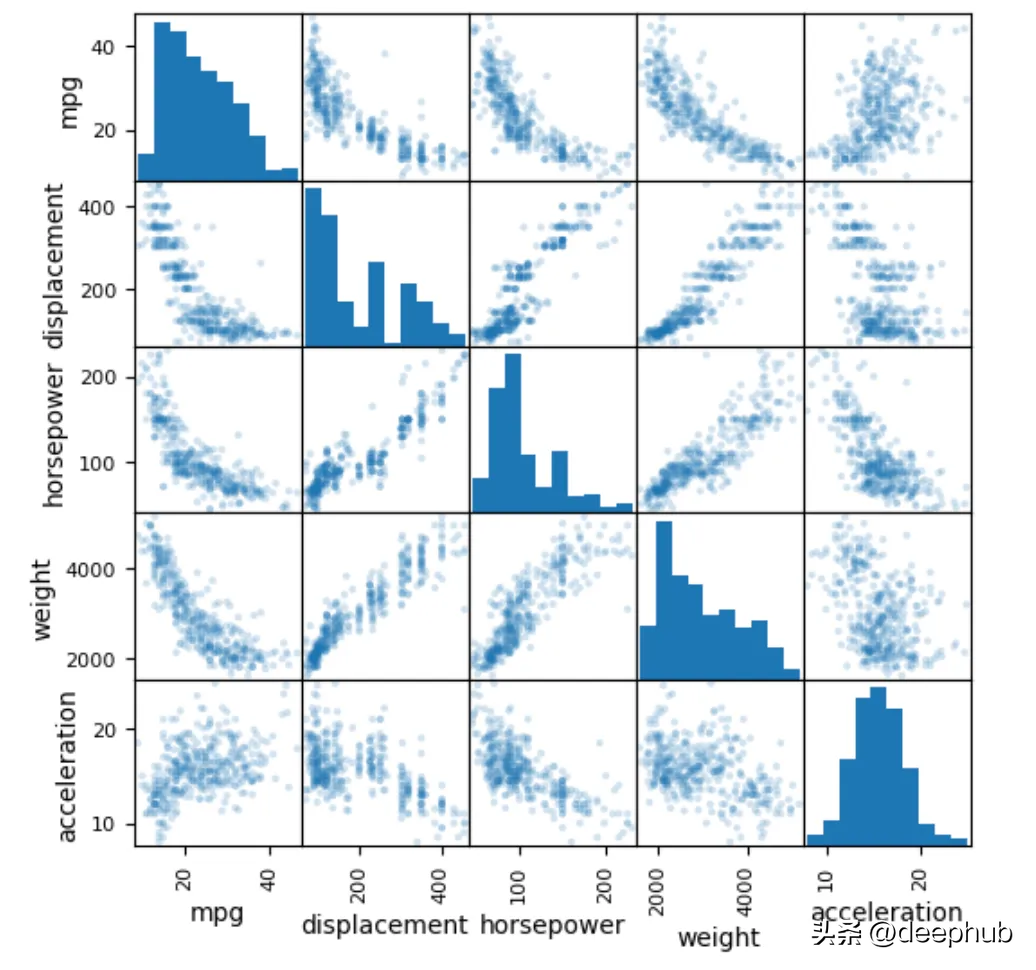
相关性的p值
如果你正在寻找一个简单的矩阵(带有p值),这是许多其他工具(SPSS, Stata, R, SAS等)默认做的,那如何在Python中获得呢?
这里就要借助科学计算的scipy库了,以下是实现的函数
from scipy.stats import pearsonr
import pandas as pd
import seaborn as sns
def corr_full(df, numeric_only=True, rows=['corr', 'p-value', 'obs']):
"""
Generates a correlation matrix with correlation coefficients,
p-values, and observation count.
Args:
- df: Input dataframe
- numeric_only (bool): Whether to consider only numeric columns for
correlation. Default is True.
- rows: Determines the information to show.
Default is ['corr', 'p-value', 'obs'].
Returns:
- formatted_table: The correlation matrix with the specified rows.
"""
# Calculate Pearson correlation coefficients
corr_matrix = df.corr(
numeric_only=numeric_only)
# Calculate the p-values using scipy's pearsonr
pvalue_matrix = df.corr(
numeric_only=numeric_only,
method=lambda x, y: pearsonr(x, y)[1])
# Calculate the non-null observation count for each column
obs_count = df.apply(lambda x: x.notnull().sum())
# Calculate observation count for each pair of columns
obs_matrix = pd.DataFrame(
index=corr_matrix.columns, columns=corr_matrix.columns)
for col1 in obs_count.index:
for col2 in obs_count.index:
obs_matrix.loc[col1, col2] = min(obs_count[col1], obs_count[col2])
# Create a multi-index dataframe to store the formatted correlations
formatted_table = pd.DataFrame(
index=pd.MultiIndex.from_product([corr_matrix.columns, rows]),
columns=corr_matrix.columns
)
# Assign values to the appropriate cells in the formatted table
for col1 in corr_matrix.columns:
for col2 in corr_matrix.columns:
if 'corr' in rows:
formatted_table.loc[
(col1, 'corr'), col2] = corr_matrix.loc[col1, col2]
if 'p-value' in rows:
# Avoid p-values for diagonal they correlate perfectly
if col1 != col2:
formatted_table.loc[
(col1, 'p-value'), col2] = f"({pvalue_matrix.loc[col1, col2]:.4f})"
if 'obs' in rows:
formatted_table.loc[
(col1, 'obs'), col2] = obs_matrix.loc[col1, col2]
return(formatted_table.fillna('')
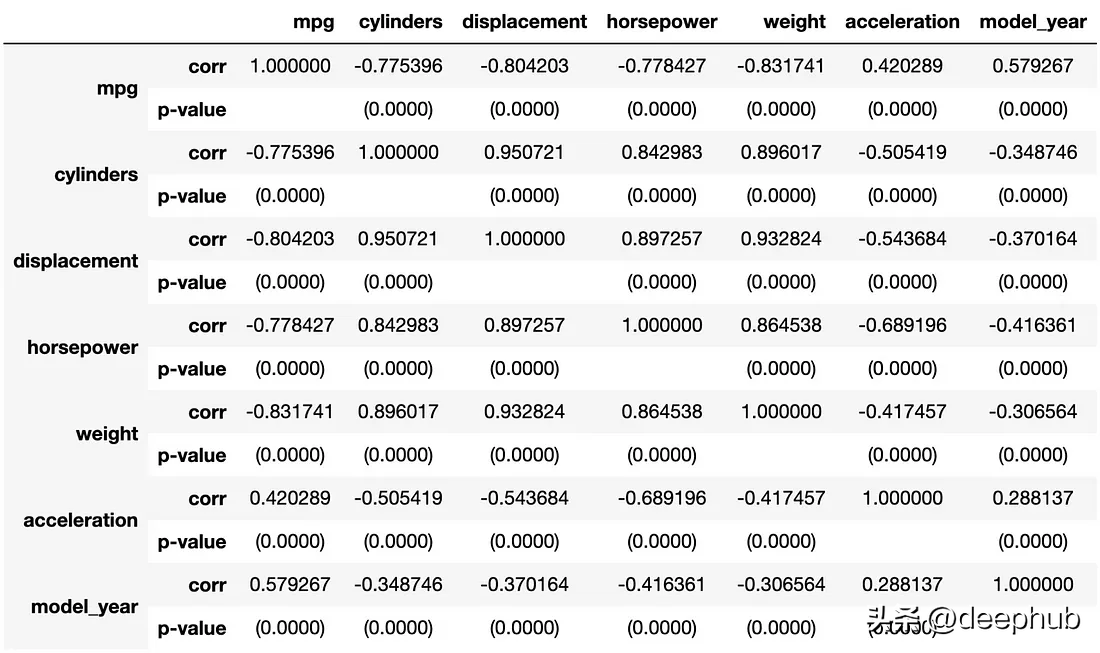
.style.set_properties(**{'text-align': 'center'}))直接调用这个函数,我们返回的结果如下:
df = sns.load_dataset('mpg')
result = corr_full(df, rows=['corr', 'p-value'])
result